1.200 Genome und 800 Transkriptome
Kontakt
Projektkoordinatoren:
Prof. Tanja Zeller
Prof. Heribert Schunkert
use.access(at)dzhk.de
Use and Access Office:
Alexandra Klatt
Referentin Klinische Forschung
Tel: 030 3465 52910
use.access(at)dzhk.de
Über das DZHKomics-Ressource-Projekt
Im Rahmen dieses Projektes wurden die Genome und Transkriptome von Probanden verschiedener populations-basierter Kohorten sequenziert und damit ein Datensatz generiert, welcher als Kontroll-Ressource für weiterführende Sequenzierungsprojekte dient. Damit trägt die DZHKomics Ressource dazu bei, unser Verständnis zu erweitern, wie Gene zum Auftreten einer Erkrankung bzw. von Erkrankungen beitragen.
Probenaufbereitung, Sequenzierungen, Prozessierungen
Im Vorfeld zu den Ganzgenom- und RNA-Sequenzierungen wurden SOPs für eine harmonisierte Probenaufbereitung eigens für die DZHKomics Ressource entwickelt. Diese Vorgehensweise sollte mögliche störende Einflüsse auf die Sequenzierungen der aus verschiedenen Kohorten stammenden Proben minimieren und eine hohe Vergleichbarkeit der generierten Sequenzdaten erreichen.
Die Vorbereitung der DNA erfolgte nach einem standardisierten Verfahren am DZHK-Standort Heidelberg, wo ebenfalls die Ganzgenomsequenzierung auf der „HiSeq-X“ Plattform der 'High Throughput Sequencing Unit' durchgeführt wurden. Die Prozessierung der Daten erfolgte in München und in Lübeck.
In Ergänzung erfolgte die Vorbereitung der RNA Proben unter standardisierten Vorgaben in München. Die RNA-Sequenzierungen wurde auf den HiSeq Sequencers in Berlin durchgeführt und deren Prozessierung anschließend in Hamburg.
Probanden
Es handelt sich um 1.300 Probanden aus sechs bevölkerungsbasierten, epidemiologischen Kohortenstudien Deutschlands. Das Geschlechterverhältnis ist fast ausgeglichen (48,3% Frauen) und das Alter bei Probenentnahme liegt im Mittel bei 57 Jahren. Es wurden Informationen zu verschiedenen kardiovaskulären Risikofaktoren erhoben, die jedoch für die meisten Probanden im Normbereich liegen. Diese Informationen sind im DZHKomics Phänotyp-Datensatz zusammengefasst, welcher die folgenden Variablen enthält:
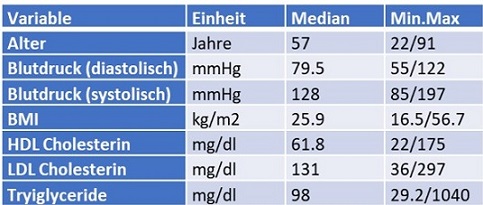
- Alter
- Geschlecht
- Größe
- Gewicht
- Blutdruck (diastolisch, systolisch)
- HDL und LDL Cholesterin
- Triglyceride
- Raucherstatus
- Diabetes
- Familiäre Fälle von Herzinfarkt
- Familiäre Fälle von Schlaganfall
- Familiäre Fälle von CHF/DCM
- Medikamente
Molekulare Daten
Die Prozessierung der Rohdaten aus der DNA-Sequenzierung erfolgte mit etablierten Pipelines und Software-Paketen (FastQC, bwa-mem und GATK) nach den Empfehlungen von GATK und gnomAD. Nach der Qualitätskontrolle sind Genotypdaten für 1099 Individuen verfügbar. Insgesamt wurden ca. 43,9 Millionen Varianten identifiziert, davon 35,8 Mio SNVs und 7 Mio InDels. Informationen zu den einzelnen Varianten inklusive Genotyphäufigkeiten stehen in unserem Genom Browser zum Durchsuchen und Herunterladen bereit.
Auch für die Prozessierung der RNA-Daten wurden etablierte Tools verwendet, u.a. FastQC, STAR, RSEM. Untransformierte und normalisierte Countdaten stehen für insgesamt 775 Individuen und 26,914 Ensembl-Gene zur Verfügung.
Kooperationspartner der DZHKomics Ressource
Beteiligte Kohorten:
Für die Generierung dieser Ressource wurden insgesamt 1.200 DNA-Proben auf Ganzgenombasis und 800 RNA-Proben (PaxGene Blood) sequenziert. Das Probenmaterial wurde dabei aus sechs an verschiedenen DZHK-Mitgliedseinrichtungen bereits existierenden epidemiologischen Kohorten-Studien ausgewählt:
- Gutenberg-Gesundheitsstudie (GHS) - Universitätsmedizin der Johannes Gutenberg-Universität Mainz
- Hamburg City Health Study (HCHS) - Universitätsklinikum Hamburg-Eppendorf
- Heidelberg Normal Kontrollen (NOKO) - Universitätsklinikum Heidelberg
- Kooperative Gesundheitsforschung in der Region Augsburg (KORA) - Helmholtz Zentrum München
- Study of Health in Pomerania (SHIP) - Universitätsmedizin Greifswald
- Institut für Epidemiologie in Kiel (IFE) - Universitätsklinikum Schleswig-Holstein

